2025-04-04
In psicologia e neuroscienze alcuni esperimenti includono delle variabili che possono variare tra soggetti, nonostante siano manipolate sperimentalmente.
Ad esempio, in psicofisica gli stimoli vengono spesso adattati ai soggetti e quindi non tutti i soggetti sono sottoposti allo stesso tipo di stimolazione.
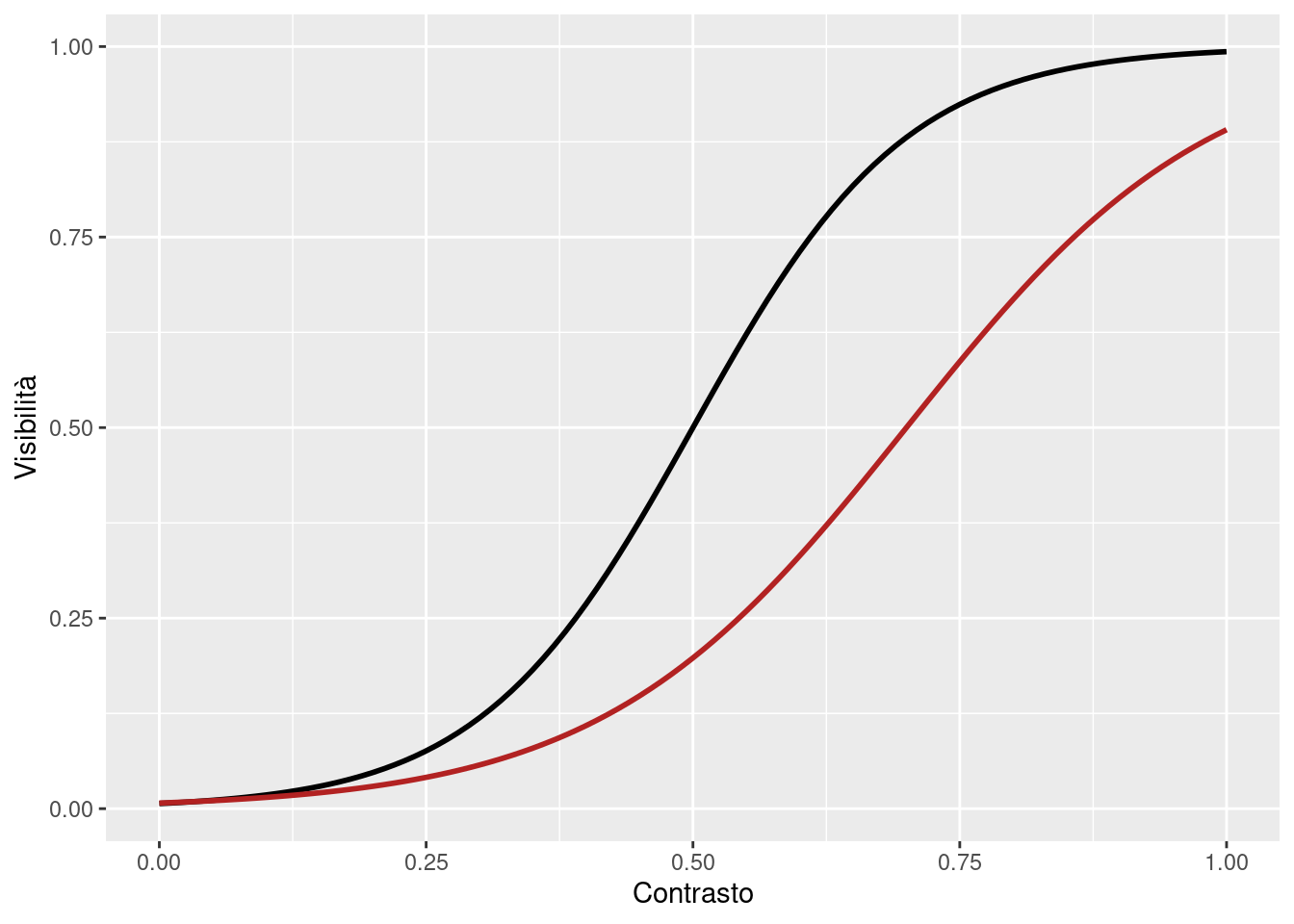
Ipotizziamo che siamo interessati a capire la sensibilità al contrasto (visibilità rispetto allo sfondo) di un gruppo di soggetti. A livello individuale possiamo ipotizzare che la relazione vera di due soggetti sia ad esempio:
Solitamente siamo interessati a stimare la soglia percettiva ed eventualmente la slope. La soglia percettiva è il livello di \(x\) (contrasto) necessario a ad avere una certa percentuale di visibilità (e.g., 50%).
Gli esperimenti possono essere tarati per adattare lo stimolo (contrasto) alle risposte permettendo di focalizzare i trial sulla parte della curva di interesse. Se proviamo a simulare due ipotetici esperimenti:
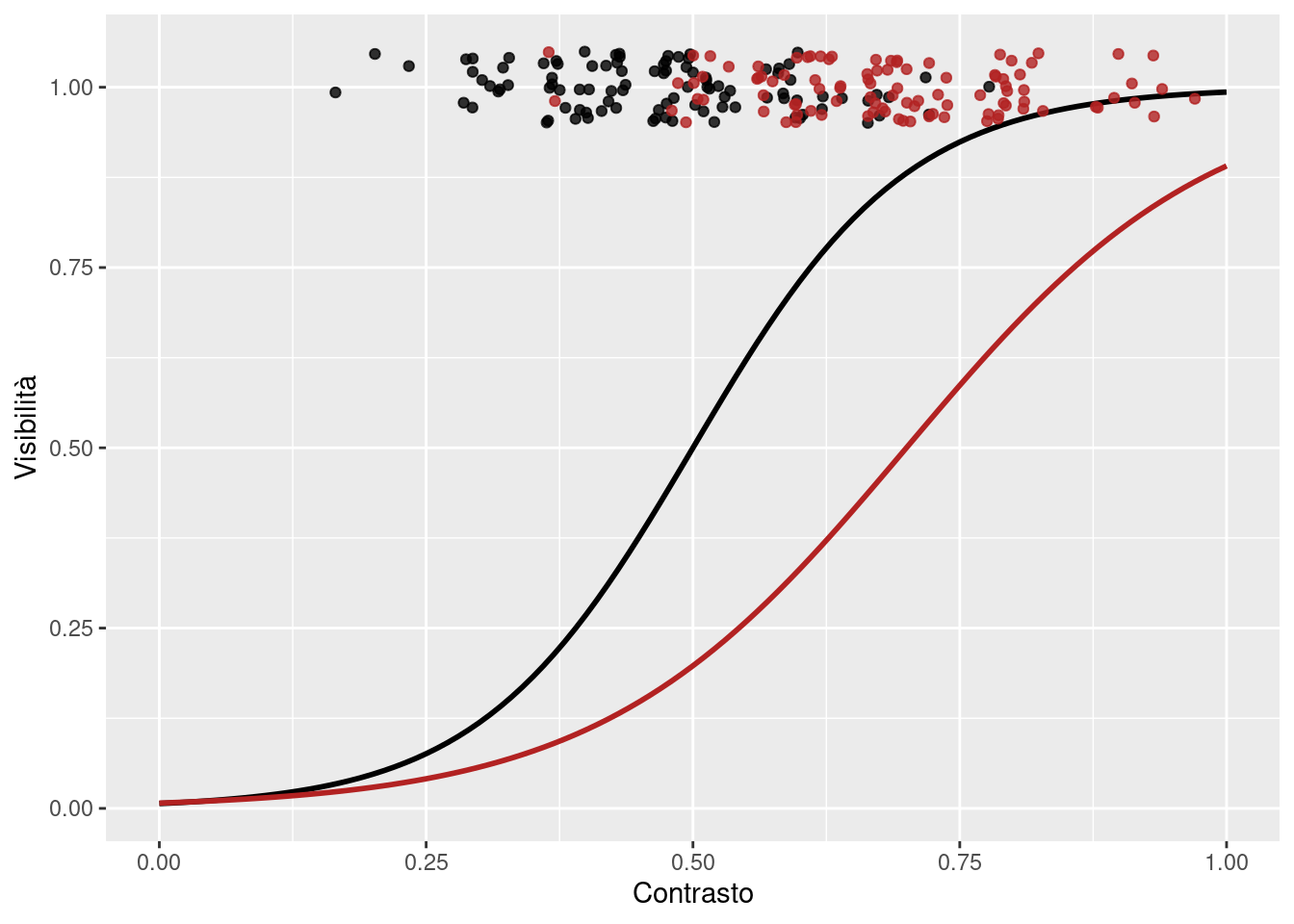
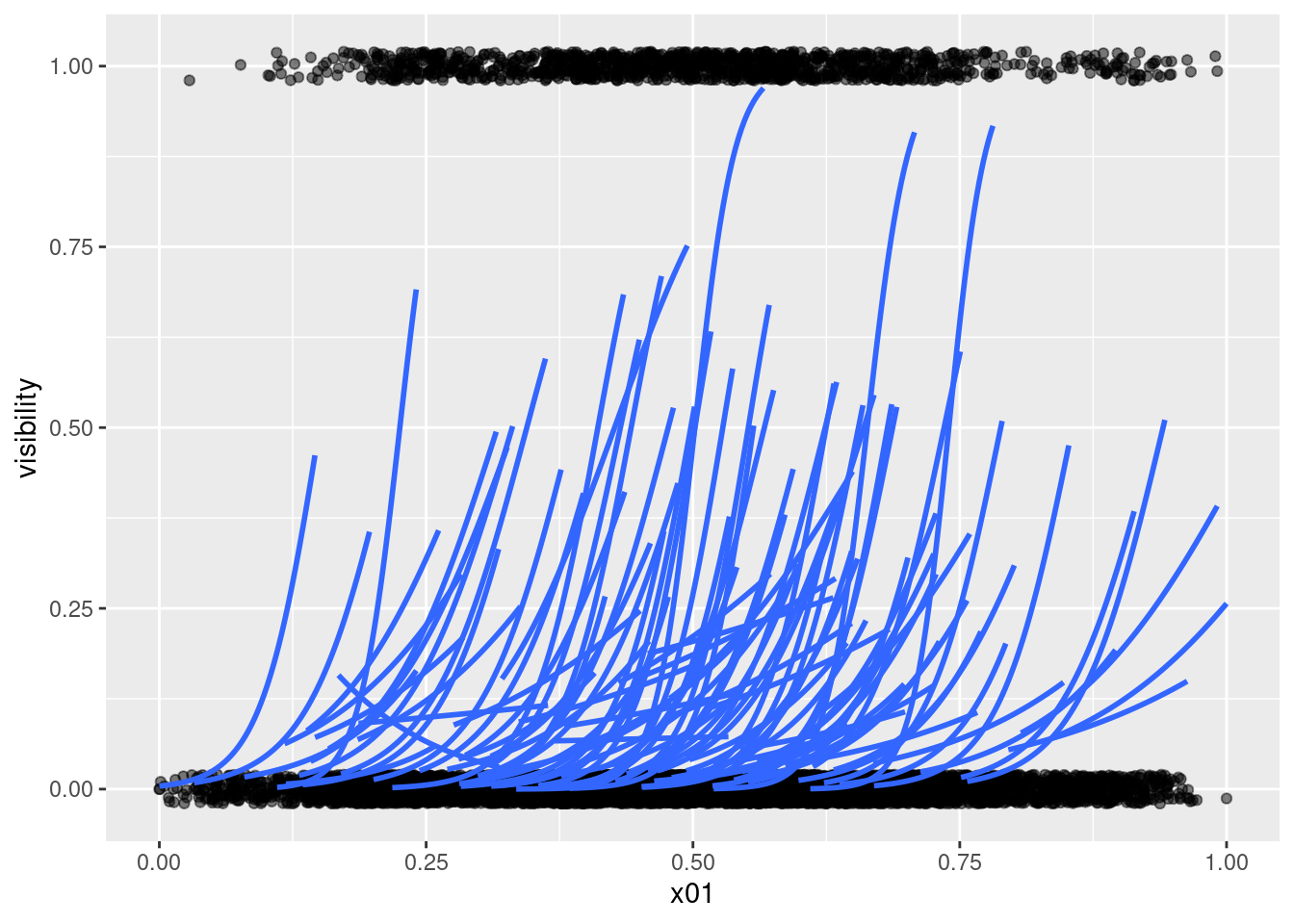
Ora carichiamo un dataset con un esperimento di questo tipo.
id trial x01 visibility
1 1 1 0.2069943 0
2 2 1 0.6339033 1
3 3 1 0.7137630 0
4 4 1 0.6853990 0
5 5 1 0.2117152 0
6 6 1 0.2450111 0Possiamo anche vedere i pattern individuali:
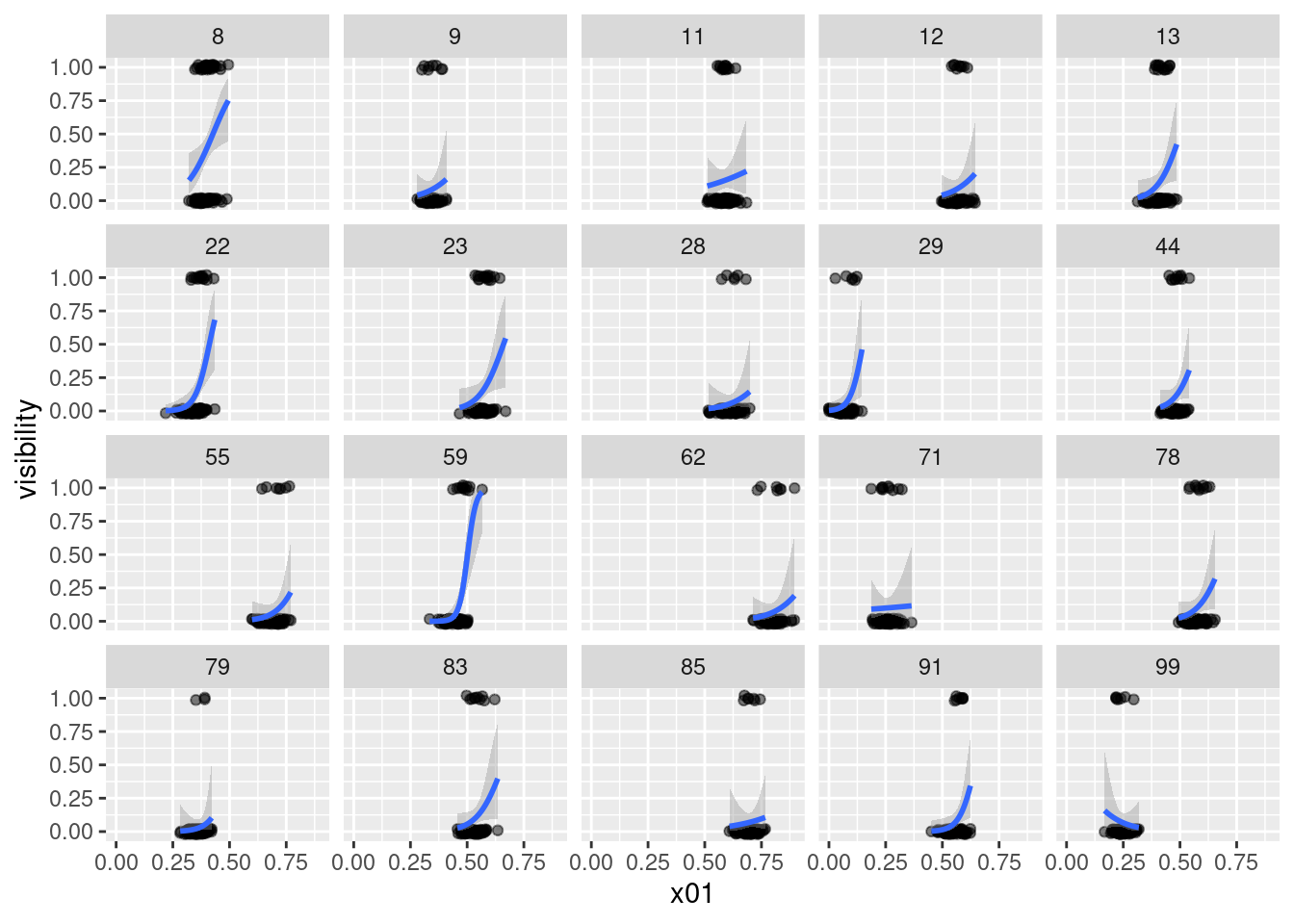
Fittiamo i nostri modelli per soggetto e vediamo il pattern di parametri:
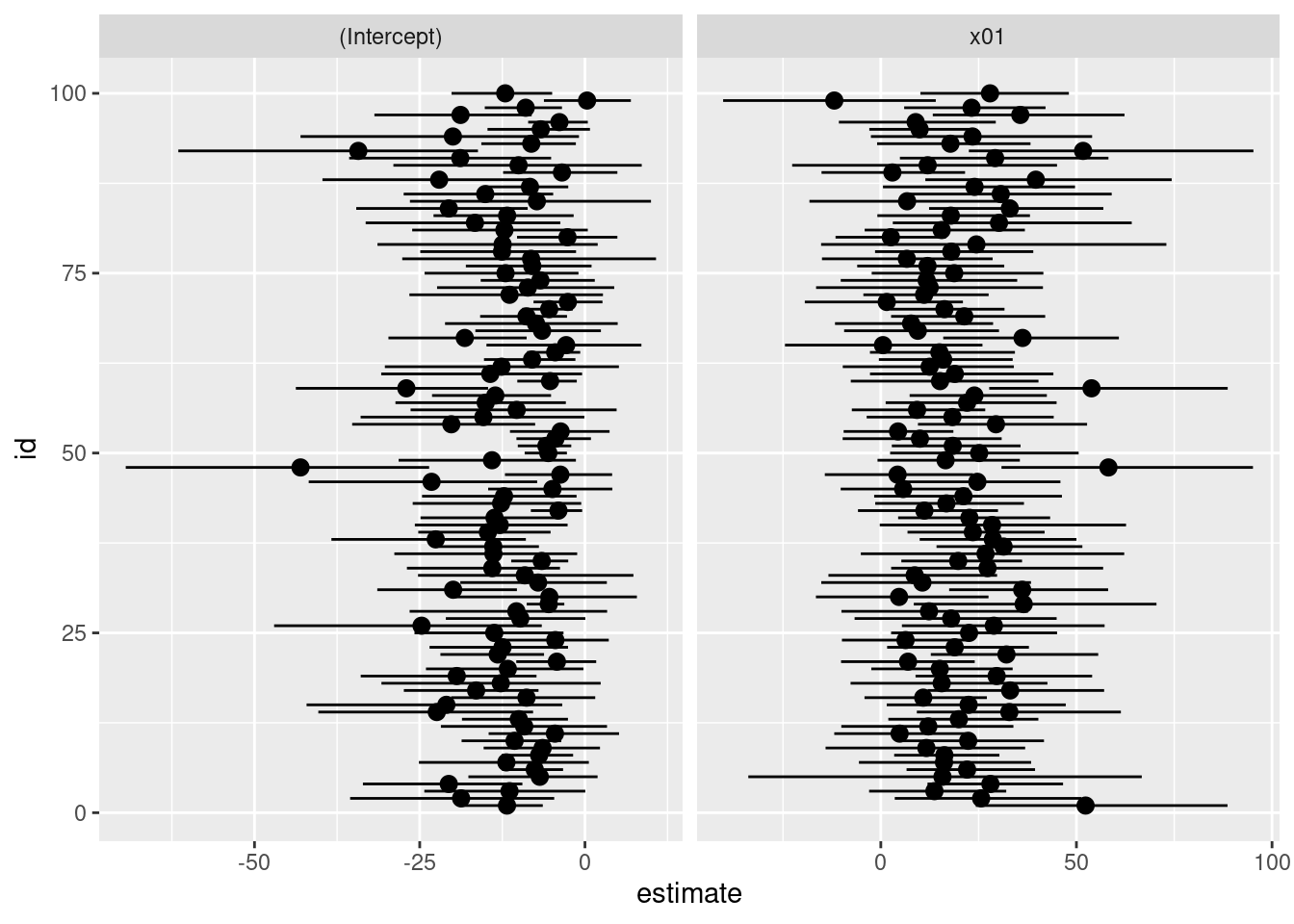
Perfetto, ora fittiamo il nostro modello:
library(lme4)
library(lmerTest)
fit <- glmer(visibility ~ x01 + (x01||id), data = dat, family = binomial(link = "logit"))
summary(fit)Generalized linear mixed model fit by maximum likelihood (Laplace
Approximation) [glmerMod]
Family: binomial ( logit )
Formula: visibility ~ x01 + (x01 || id)
Data: dat
AIC BIC logLik -2*log(L) df.resid
7387.3 7416.2 -3689.7 7379.3 9996
Scaled residuals:
Min 1Q Median 3Q Max
-1.2114 -0.4091 -0.3261 -0.2499 8.6903
Random effects:
Groups Name Variance Std.Dev.
id (Intercept) 2.217 1.489
id.1 x01 1.896 1.377
Number of obs: 10000, groups: id, 100
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -6.342 1.013 -6.258 3.91e-10 ***
x01 8.738 2.017 4.333 1.47e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Correlation of Fixed Effects:
(Intr)
x01 -0.984library(ggeffects)
plot(ggeffect(fit))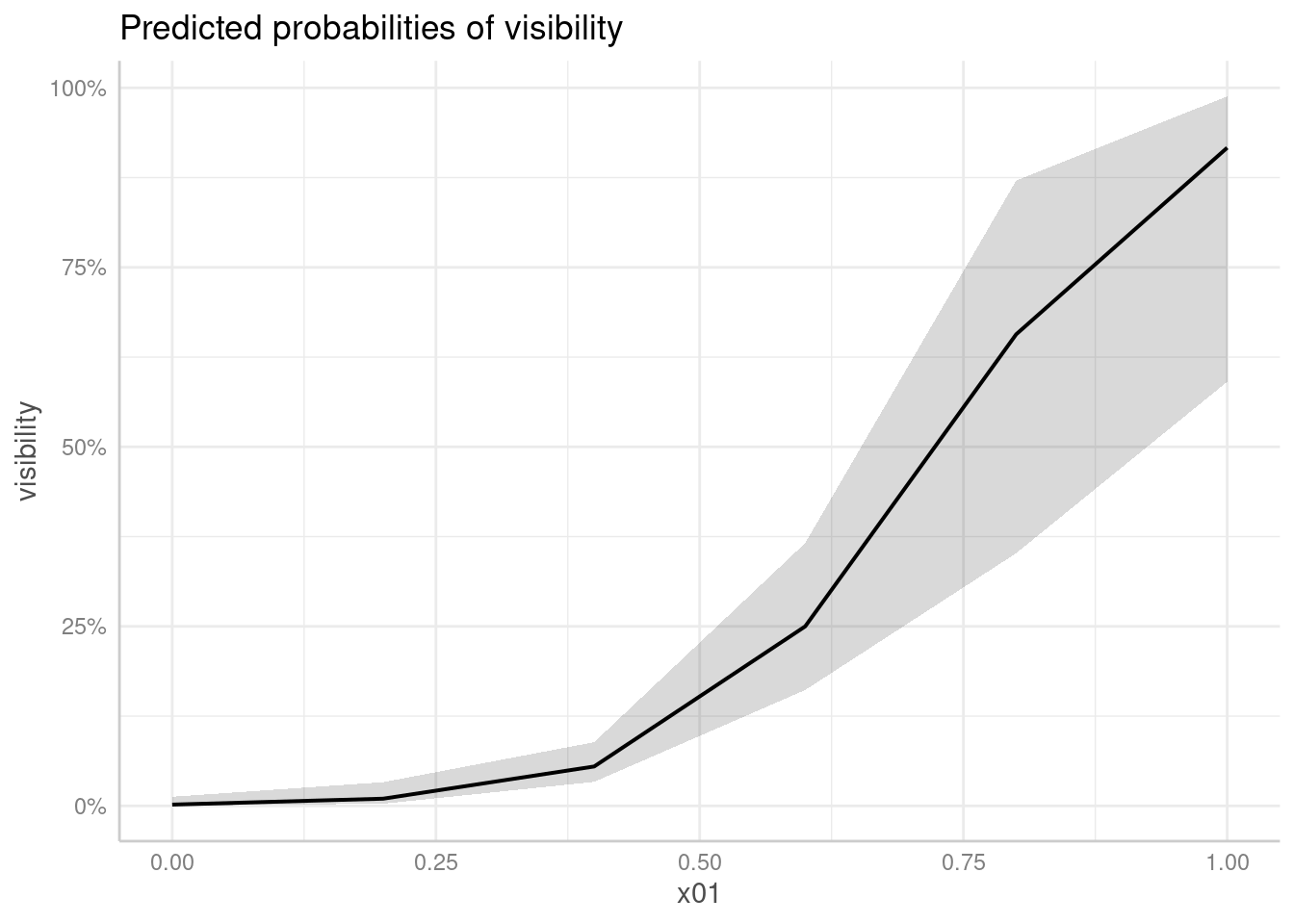
L’effetto fisso stimato dal modello è 8.74. Al netto dello shrinkage, questo è decisamente inferiore rispetto alla media delle slope stimate sui singoli soggetti. Quale potrebbe essere il motivo?
Il problema principale è che abbiamo un effetto del cluster (soggetti) sulla nostra variabile \(x\) (vi dice qualcosa il Simpson’s paradox?). Ogni soggetto ha il suo esperimento con un incremento chiaro (e consistente) della visibilità in funzione del contrasto MA i livelli di contrasto di ogni soggetto sono diversi uno dall’altro.
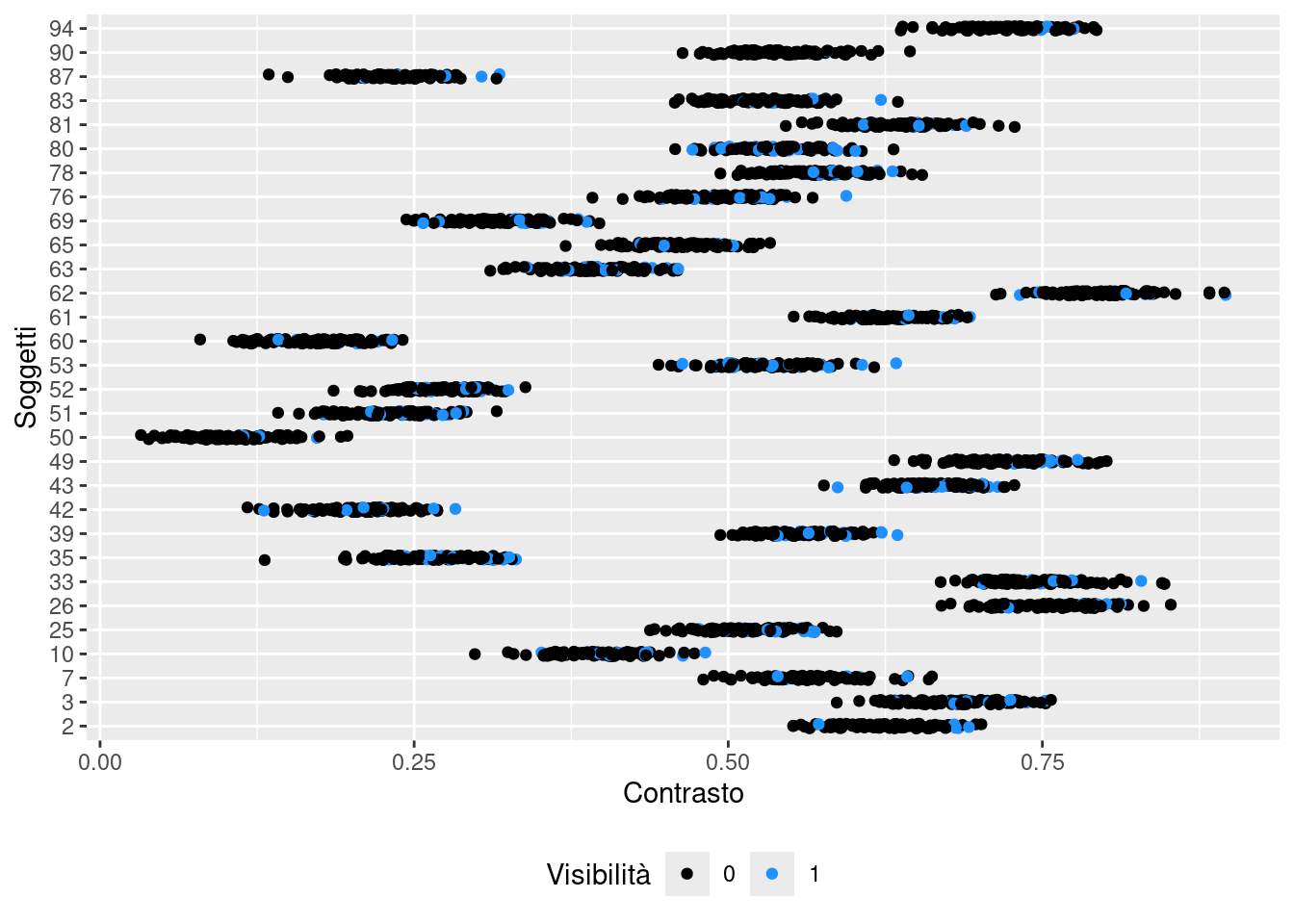
Quindi in realtà non stiamo stimando esattamente quello che vorremmo, ovvero l’effetto medio (e la sua variabilità) del contrasto in ogni soggetto. Idealmente dovremmo calcolarlo su ogni soggetto e poi fare la media.
Per fare questo è necessario trasformare la \(x\), centrando i valori di contrasto sulla media di ogni soggetto.
cmc <- function(x, cluster){
x - cm(x, cluster)
}
cm <- function(x, cluster){
cmm <- tapply(x, cluster, mean)
cmn <- tapply(x, cluster, length)
cmm[as.character(cluster)]
}
dat_ex$x01_cmc <- cmc(dat_ex$x01, dat_ex$id)
fit_cmc <- glmer(visibility ~ x01_cmc + (x01_cmc||id), data = dat_ex, family = binomial(link = "logit"))
summary(fit_cmc)Generalized linear mixed model fit by maximum likelihood (Laplace
Approximation) [glmerMod]
Family: binomial ( logit )
Formula: visibility ~ x01_cmc + (x01_cmc || id)
Data: dat_ex
AIC BIC logLik -2*log(L) df.resid
7081.7 7110.5 -3536.8 7073.7 9996
Scaled residuals:
Min 1Q Median 3Q Max
-1.5490 -0.4042 -0.3101 -0.2293 6.2565
Random effects:
Groups Name Variance Std.Dev.
id (Intercept) 0.2294 0.4789
id.1 x01_cmc 7.6120 2.7590
Number of obs: 10000, groups: id, 100
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -2.15515 0.05998 -35.93 <2e-16 ***
x01_cmc 18.17924 1.03817 17.51 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Correlation of Fixed Effects:
(Intr)
x01_cmc -0.224Se erroneamente (qualche volta viene fatto), decidiamo di aggregare i dati a livello del soggetto. Possiamo nel migliore dei casi attenuare o distorcere la stima oppure stimare un pattern anche opposto.
dat_agg <- dat_ex |>
group_by(id) |>
summarise(p = mean(visibility),
n = n(),
x01 = mean(x01))
head(dat_agg)# A tibble: 6 × 4
id p n x01
<int> <dbl> <int> <dbl>
1 1 0.11 100 0.171
2 2 0.08 100 0.623
3 3 0.12 100 0.680
4 4 0.21 100 0.679
5 5 0.02 100 0.180
6 6 0.17 100 0.264fit_agg <- glmer(p ~ x01 + (1|id), data = dat_agg, weights = n, family = binomial(link = "logit"))
summary(fit_agg)Generalized linear mixed model fit by maximum likelihood (Laplace
Approximation) [glmerMod]
Family: binomial ( logit )
Formula: p ~ x01 + (1 | id)
Data: dat_agg
Weights: n
AIC BIC logLik -2*log(L) df.resid
633.0 640.8 -313.5 627.0 97
Scaled residuals:
Min 1Q Median 3Q Max
-1.62242 -0.44308 0.02905 0.35862 1.33912
Random effects:
Groups Name Variance Std.Dev.
id (Intercept) 0.2064 0.4543
Number of obs: 100, groups: id, 100
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -1.9989 0.1605 -12.455 <2e-16 ***
x01 -0.0477 0.3052 -0.156 0.876
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Correlation of Fixed Effects:
(Intr)
x01 -0.938